Составляем слова из букв
Получается, читать ДНК нелегко. Через 15 лет после открытия пространственной структуры ДНК Рэй Ву и Дейл Кайзер сумели распознать 12 нуклеотидов в геноме вируса[49]. Весь этот геном содержит около 48 тысяч нуклеотидов. Пять лет спустя Аллан Максам и Уолтер Гилберт получили последовательность из 24 нуклеотидов ДНК – область lac-оператора (см. главу 4). Для этого потребовалось два года напряженной работы – на секвенирование с такой скоростью генома человека ушло бы 250 миллионов лет. Явно были нужны куда более эффективные методы.
И такие методы появились1. Несколько следующих страниц мы посвятим именно им – и не только из-за их значимости в современном мире, но и потому, что одним своим существованием они заявляют о важности познания физической природы ДНК и других биомолекул. Понятийным фоном для этой практической главы послужат такие свойства, как размер, жесткость и электрический заряд, и такие темы, как самосборка и предсказуемая случайность.
В 1976–1977 годах появились два остроумных метода определения последовательности ДНК: один разработали те самые Максам и Гилберт, а другой – Фредерик Сэнгер с коллегами. Метод Сэнгера оказался проще и в итоге доминировал в отрасли больше двух десятилетий. Я начну рассказ о техниках чтения ДНК именно с секвенирования по Сэнгеру.
Представьте, что у вас нет возможности последовательно, буква за буквой читать слово М-О-Л-Е-К-У-Л-А, но вы видите перед собой множество оборванных копий этого слова, в каждой из которых различима лишь последняя буква: «??Л», «?О», «?????У» и так далее. Заметив, что все трехбуквенные фрагменты оканчиваются на Л, все четырехбуквенные – на Е и так далее, вы установите, что вместе они образуют слово МОЛЕКУЛА. В принципе, в этом и состоит суть метода Сэнгера и еще нескольких подходов: чтение осуществляется путем распознавания отдельных кусочков, а не последовательного движения по цепи.
В главе 1 мы уже описывали несколько шагов из этого рецепта. Представьте себе не весь геном, а что-то более податливое – скажем, фрагмент ДНК размером несколько сотен пар нуклеотидов. Полимеразная цепная реакция (ПЦР) создает несметное число копий этого фрагмента, а тепло разделяет каждую двойную спираль на одноцепочечные половинки. Пока забудьте об одной из них и представьте миллионы идентичных одноцепочечных ДНК. Как вы помните, ДНК-полимераза создает идеальный комплемент любой цепи ДНК: руководствуясь принципом комплементарности, она сшивает из свободных нуклеотидов новую партнерскую цепь. А теперь представьте, что ученый применяет ДНК-полимеразу для репликации ДНК, как в нормальной ПЦР, но вводит в массив свободных нуклеотидов небольшое количество дефектных молекул: они мало отличаются от обычных A, Ц, Г, T и по-прежнему встраиваются в растущую цепь ДНК, но уже к ним новые нуклеотиды присоединиться не могут. Полимераза не умеет отбраковывать такое строительное сырье, и если ей попадается нормальный свободный нуклеотид, новая цепь удлиняется, если же встраивается измененный вариант, он блокирует дальнейший синтез и остается в цепи последним. Поскольку добавление терминирующих нуклеотидов происходит по воле случая и сравнительно редко, ученый получает множество цепочек ДНК, которые начинаются одинаково, но различаются по длине.
Пока складывается впечатление, что измененные нуклеотиды просто нарушают ход ПЦР, однако они сконструированы так, чтобы не только препятствовать удлинению ДНК, но и работать «маячками» – испускать свет одного из четырех цветов, уникальных для не-совсем-A, не-совсем-Ц, не-совсем-Г и не-совсем-T. Распознаваемым сигналом могут служить не только цвета. Сначала в секвенировании по Сэнгеру использовали радиоактивные метки, а вместо ПЦР (которую тогда еще не изобрели) для клонирования ДНК привлекали бактерий. Здесь мы описываем более поздние и эффективные разновидности метода, основанные, однако, на тех же принципах[50].
На последнем этапе плавления ДНК-дуплекс разделяется на две нити, и фрагменты разной длины оказываются маркированными на концах: теперь они напоминают те куски слов, где видно лишь последнюю букву. Ученый по-прежнему не знает длину конкретных фрагментов, и они слишком малы, чтобы наблюдать их в видимом свете. Как мы узнали из главы 1, ПЦР эксплуатирует одну из важных физических характеристик ДНК – плавление, то есть разделение двойной спирали на отдельные цепи при превышении специфической температуры. Секвенирование по Сэнгеру задействует и другое важное свойство ДНК – электрический заряд.
Как мы отметили в главе 3, описывая наматывание ДНК на гистоны, ДНК заряжена отрицательно, а значит, ее можно перемещать с помощью электрических полей: притягивать к положительно заряженным электродам и отталкивать от отрицательно заряженных2. В обычной воде кусочки ДНК движутся со сходной скоростью вне зависимости от размера. У крупных фрагментов заряд больше, а следовательно, их толкает бо́льшая электрическая сила, но и сопротивление жидкости они встречают бо́льшее. Физика масштабирования этих двух сил в зависимости от размера фрагмента сложна и неочевидна, но в итоге их эффекты почти нивелируют друг друга, и в негустой жидкости подвижность фрагмента слабо зависит от его длины. А вот в гелевых пластинах ситуация меняется. Длинные молекулярные цепочки пищевого желатина, например, спутываются и формируют пористую трехмерную сеть, пропускающую воду. Чтобы перемещаться по гелю, однонитевая ДНК (черная на рисунке) должна змейкой пробираться через поры – по-научному это называется рептацией. ДНК приходится то и дело извиваться, с чем короткие молекулы справляются гораздо быстрее, чем длинные.
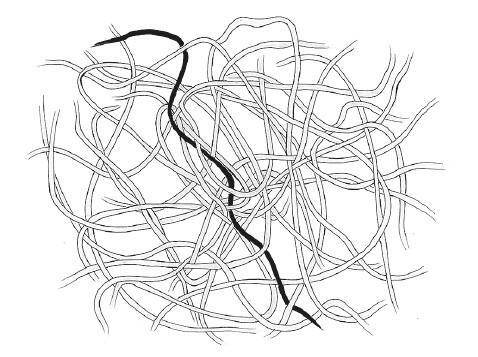
Предсказуемая случайность броуновского движения – важнейшее условие для такого способа перемещения. Без нее ДНК застревала бы в геле, каким бы сильным ни было электрическое поле: попади концы нити в разные поры, молекула повисла бы на препятствии, как полотенце на веревке, и не смогла бы высвободиться. Но благодаря броуновскому движению ДНК постоянно колеблется и переориентируется, выбираясь из одного отверстия и проскальзывая сквозь другое. Статистическая прогнозируемость микроскопической случайности дает нам четко определенную и поддающуюся математической обработке скорость движения молекулы.
Итак, после амплификации ДНК с проставлением концевых меток и прогона фрагментов ДНК через гель под действием электрического поля[51] мы получаем возможность прочитать нуклеотидную последовательность. Все фрагменты одной и той же длины будут светиться одним цветом. Допустим, нити из 27 нуклеотидов заканчиваются модифицированным Ц, несущим, скажем, красную метку. А нити из 28 нуклеотидов заканчиваются модифицированным T с синей меткой. И так далее. Среди нитей из 27 нуклеотидов нет ни одной синей, поскольку все фрагменты этой длины, как точные копии друг друга, должны заканчиваться Ц, а все терминирующие Ц – красные. (Быть может, читая последние страницы, вы переживали за выпавшую из нашего поля зрения вторую исходную цепь ДНК, которая могла бы стать матрицей для второго набора молекулярных фрагментов. Не бойтесь: в секвенировании по Сэнгеру особый подбор праймеров заставляет ДНК-полимеразу работать только с одной из цепей двойной спирали, потому вторая вообще не реплицируется.)
Все фрагменты ДНК совместно попадают в тонкую трубку с гелем, проходя по которой, разделяются из-за разной скорости движения. Наблюдая за флуоресценцией проходящих через трубку точек, ученый фиксирует, например, вначале красный сигнал, затем синий, еще один синий, за ним зеленый и так далее, и интерпретирует их как последовательность ЦTTA+++++. Вот мы и прочитали ДНК.